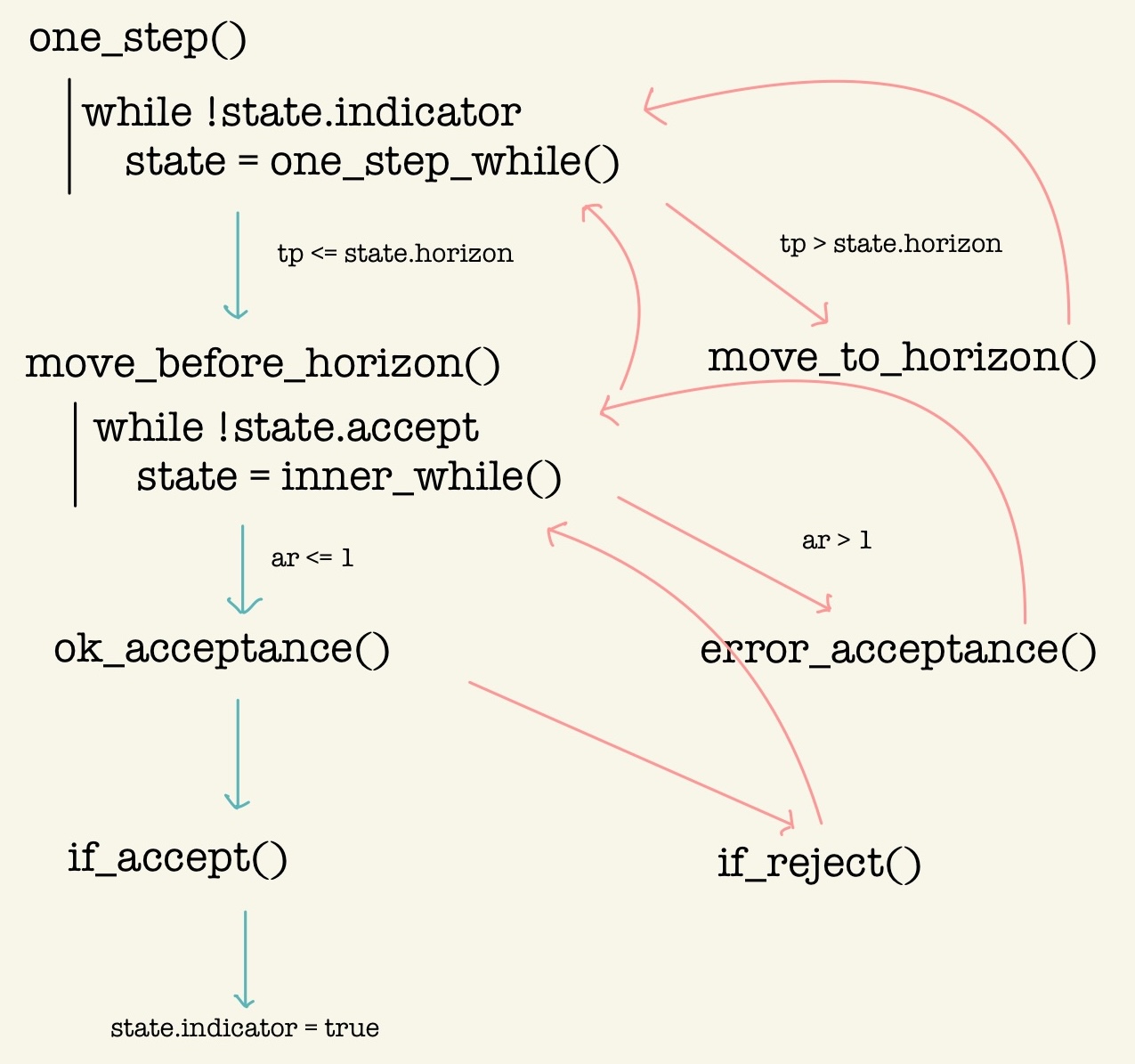
graph TD;
A["one_step()"] -->|state.indicator=false| B["one_step_while()"]
B --> C{tp <= state.horizon}
C -->|No| D[move_to_horizon]
C -->|Yes| E[move_before_horizon]
E -->|state.accept=false| G[inner_while]
G --> I{ar <= 1.0}
I -->|No| J[error_acceptance]
I -->|Yes| K[ok_acceptance]
K -->|reject| L["if_reject()"]
L -->|場合によっては move_to_horizon2| N["inner_while() か one_step_while() まで戻る"]
K -->|accept| O["if_accept()
state.indicator=true
"]
D --> B
J -->|horizon を縮める| N
1 速習 PDMP
1.1 はじめに
PDMP (Piecewise Deterministic Markov Process) または 連続時間 MCMC とは,名前の通り MCMC のサンプリングを連続時間で行うアルゴリズムである:

詳しくは次の記事も参照:
A Blog Entry on Bayesian Computation by an Applied Mathematician
$$
$$
1.2 使い方
using PDMPFlux2 pdmp_jax パッケージ
本節では PDMP のシミュレーションに自動微分を応用した (Andral and Kamatani, 2024) のアルゴリズムを紹介する.
2.1 デモ
(Neal, 2003) が slice sampling のデモ用に定義した 漏斗分布 を考える: \[ p(y,x)=\phi(y;0,3)\prod_{i=1}^9\phi(x_i;0,e^{y/2}),\qquad y\in\mathbb{R},x\in\mathbb{R}^9. \]
import jax
from jax.scipy.stats import multivariate_normal
from jax.scipy.stats import norm
import jax.numpy as jnp
def funnel(d=10, sig=3, clip_y=11):
"""Funnel distribution for testing. Returns energy and sample functions."""
def unbatched(x):
y = x[0]
log_density_y = - y**2 / 6
variance_other = jnp.exp(y/2)
log_density_other = - jnp.sum(x[1:]**2) / (2 * variance_other)
return - log_density_y - log_density_other
def sample_data(n_samples):
# sample from Nd funnel distribution
y = (sig * jnp.array(np.random.randn(n_samples, 1))).clip(-clip_y, clip_y)
x = jnp.array(np.random.randn(n_samples, d - 1)) * jnp.exp(-y / 2)
return jnp.concatenate((y, x), axis=1)
return unbatched, sample_dataimport pdmp_jax as pdmp
dim = 10
U, _ = funnel(d=dim)
grad_U = jax.grad(U)
seed = 8
xinit = jnp.ones((dim,)) # initial position
vinit = jnp.ones((dim,)) # initial velocity
grid_size = 0
N_sk = 100000 # number of skeleton points
N = 100000 # number of samples
sampler = pdmp.ZigZag(dim, grad_U, grid_size)
# sample the skeleton of the process
out = sampler.sample_skeleton(N_sk, xinit, vinit, seed, verbose = True) # takes only 3 seconds on my M1 Mac
# sample from the skeleton
sample = sampler.sample_from_skeleton(N,out)import seaborn as sns
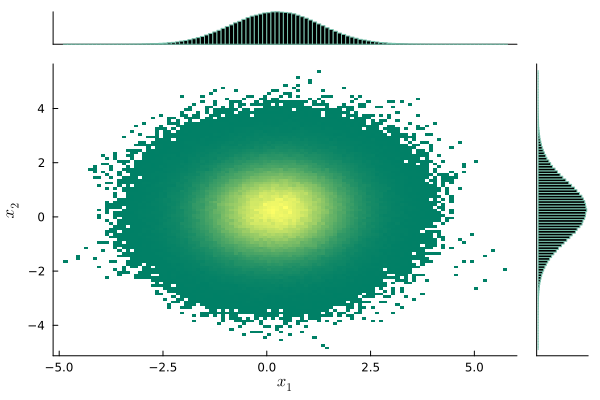
sns.jointplot(x = sample[:,0],y = sample[:,1])
plt.show()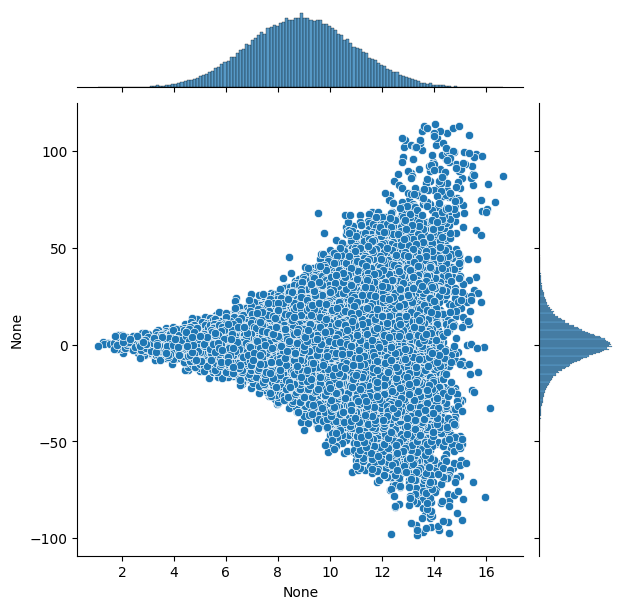
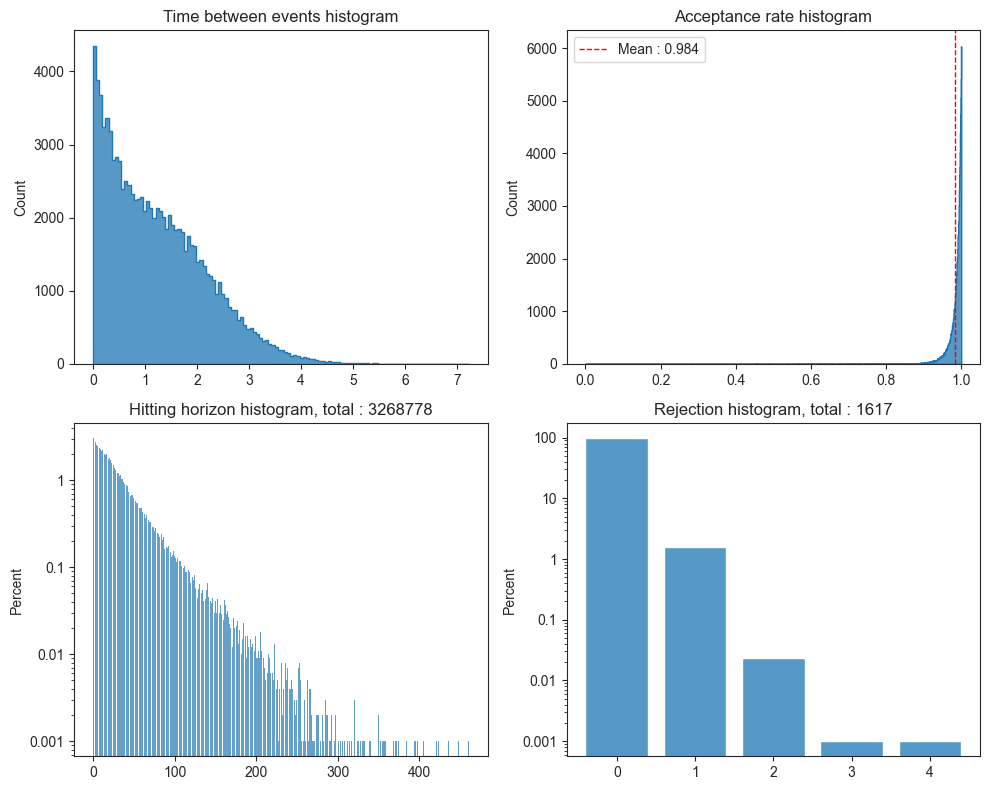
number of error bound : 46817
2.2 サンプリングループの構造
ar=lambda_t/state.lambda_barによって Poisson thinning を行う.ただし,
lambda_barとは近似的な上界であり,「最も近い直前の grid 上の点での値」でしかない.当然lambda_tを超過し得る.そのような場合にerror_acceptance()に入る.error_acceptance()に入った場合,horizonを縮めてより慎重に同じ区間を Poisson thinning しなおす.adaptive=trueの場合はこのタイミングでhorizonを恒久的に縮める.最後
if_reject()に入った場合,horizonに到達したらone_step_while()まで戻るが,そうでない場合はinner_while()まで戻る実装がなされている.
2.3 適応的なステップサイズ
3 PDMPFlux.jl パッケージ
3.1 デモ
using PDMPFlux
using Random, Distributions, Plots, LaTeXStrings, Zygote, LinearAlgebra
"""
Funnel distribution for testing. Returns energy and sample functions.
For reference, see Neal, R. M. (2003). Slice sampling. The Annals of Statistics, 31(3), 705–767.
"""
function funnel(d::Int=10, σ::Float64=3.0, clip_y::Int=11)
function neg_energy(x::Vector{Float64})
v = x[1]
log_density_v = logpdf(Normal(0.0, 3.0), v)
variance_other = exp(v)
other_dim = d - 1
cov_other = I * variance_other
mean_other = zeros(other_dim)
log_density_other = logpdf(MvNormal(mean_other, cov_other), x[2:end])
return - log_density_v - log_density_other
end
function sample_data(n_samples::Int)
# sample from Nd funnel distribution
y = clamp.(σ * randn(n_samples, 1), -clip_y, clip_y)
x = randn(n_samples, d - 1) .* exp.(-y / 2)
return hcat(y, x)
end
return neg_energy, sample_data
end
function plot_funnel(d::Int=10, n_samples::Int=10000)
_, sample_data = funnel(d)
data = sample_data(n_samples)
# 最初の2次元を抽出(yとx1)
y = data[:, 1]
x1 = data[:, 2]
# 散布図をプロット
scatter(y, x1, alpha=0.5, markersize=1, xlabel=L"y", ylabel=L"x_1",
title="Funnel Distribution (First Two Dimensions' Ground Truth)", grid=true, legend=false, color="#78C2AD")
# xlim と ylim を追加
xlims!(-8, 8) # x軸の範囲を -8 から 8 に設定
ylims!(-7, 7) # y軸の範囲を -7 から 7 に設定
end
plot_funnel()
function run_ZigZag_on_funnel(N_sk::Int=100_000, N::Int=100_000, d::Int=10, verbose::Bool=false)
U, _ = funnel(d)
grad_U(x::Vector{Float64}) = gradient(U, x)[1]
xinit = ones(d)
vinit = ones(d)
seed = 2024
grid_size = 0 # constant bounds
sampler = ZigZag(d, grad_U, grid_size=grid_size)
out = sample_skeleton(sampler, N_sk, xinit, vinit, seed=seed, verbose = verbose)
samples = sample_from_skeleton(sampler, N, out)
return out, samples
end
output, samples = run_ZigZag_on_funnel() # 4分かかる
jointplot(samples)
このデモコードは Zygote.jl による自動微分を用いると 5:29 かかっていたところが,ForwardDiff.jl による自動微分を用いると 0:21 に短縮された.
3.2 Zygote.jl と ForwardDiff.jl による自動微分
Zygote.jl は FluxML が開発する Julia の自動微分パッケージである.
using Zygote
@time Zygote.gradient(x -> 3x^2 + 2x + 1, 5) 0.830714 seconds (3.45 M allocations: 168.328 MiB, 3.95% gc time, 99.98% compilation time)(32.0,)f(x::Vector{Float64}) = 3x[1]^2 + 2x[2] + 1
g(x) = Zygote.gradient(f,x)
g([1.0,2.0])([6.0, 2.0],)大変柔軟な実装を持っており,広い Julia 関数を微分できる.
ForwardDiff.jl (Revels et al., 2016) は Zygote.jl よりも高速な自動微分を特徴としている.
using ForwardDiff
@time ForwardDiff.derivative(x -> 3x^2 + 2x + 1, 5) 0.068804 seconds (260.59 k allocations: 12.725 MiB, 16.89% gc time, 99.92% compilation time)323.3 Brent の最適化
Optim.jl は Julia の最適化パッケージであり,デフォルトで Brent の最適化アルゴリズムを提供する.
using Optim
f(x) = (x-1)^2
result = optimize(f, 0.0, 1.0)
result.minimizer0.9999999849478423.4 StatsPlots.jl による可視化
StatsPlots は現在 Plots.jl に統合されている.
また PDMPFlux.jl は marginalhist を wrap した jointplot() 関数を提供する.
3.5 ProgressBars.jl による進捗表示
ProgressBars.jl は tqdm の Julia wrapper を提供する.PDMPFlux.jl ではこちらを採用して,サンプリングの実行進捗を表示する.
なお ProgressMeter.jl も同様の機能を提供しており,有名な別の PDMP パッケージである ZigZagBoomerang.jl ではこちらを採用している.
4 終わりに
今後の確率的プログラミングの1つの焦点は自動微分かもしれない.
今回のパッケージ開発で,少なくとも v0.2.0 の時点では,プログラムに与える U_grad は多くの場合(10 次元の多変量 Gauss,50 次元の Banana など) Zygote.jl が少し速い(Funnel 分布では ForwardDiff.jl が速い).
しかし上界を構成する際の func の微分は ForwardDiff.jl の方が圧倒的に速い.大変に不可思議である.
だから現在の実装は Zygote.jl と ForwardDiff.jl の両方を用いている.
5 ToDo
Zig-Zag 以外のサンプラーの実装
ZigZag(dim) は自動で知ってほしい
Try clause 内の else を用いているので Julia 1.8 以上が必要.
MCMCChains のような plot.jl を完成させる.
PDMPFlux.jlのドキュメントを整備する.ZigZagBoomerang.jlを見習って統合したり API をつけたり?Turing エコシステムと統合できたりしないか?
Rngを指定できるようにする?pdmp-jaxでは 37 秒前後かかる Banana density の例が,PDMPFlux.jlでは 2 分前後かかる.- しかし,Julia の方が数値誤差が少ないのか,banana potential の対称性がうまく結果に出る.尾が消えたりしない.
- →
ForwardDiff.jlを採用したところ,02:05 から 10 分以上に変化した.ReverseDiff.jlを採用したところ 4:44 になった.50 次元というのが微妙なところなのかもしれない.
Funnel 分布で試したところ,
PDMPFlux.jlの棄却率が極めて高い.